
近日,生命科学学院廖明帜教授团队在染色质环预测及三维基因组研究方面取得新进展,研究成果以“CLAMP: predicting specific protein-mediated chromatin loops in diverse species with a chromatin accessibility language model”为题发表在《Genome Biology》。生命科学学院在读硕士研究生何志杰、军事医学研究院助理研究员孙昱及副研究员李昊为论文共同第一作者,生命科学学院廖明帜教授、军事医学科学院伯晓晨研究员和陈河兵副研究员为共同通讯作者。
真核生物的基因表达调控高度依赖于其基因组的三维空间组织结构,其中染色质环作为连接远端调控元件(如增强子与启动子)的关键结构基础,发挥着核心作用。然而,当前广泛使用的高通量实验技术(如Hi-C、ChIA-PET等)受限于较高的成本和有限的分辨率,难以在多种生物学场景中实现对染色质环的大规模鉴定。与此同时,现有的计算预测方法大多局限于特定物种或蛋白质,且往往缺乏足够的生物学可解释性。
为解决上述挑战,研究团队提出了名为CLAMP的全新深度学习框架。该框架创新性地利用大规模跨物种染色质可及性数据对DNA语言模型进行预训练,不仅捕获了基因组序列的深层语法特征,还整合了局部表观遗传信号和基因组空间距离等信息。基于这一染色质可及性语言模型,CLAMP实现了跨物种、跨细胞类型的染色质环高精度预测,并进一步揭示了三维基因组折叠背后的潜在调控规律。
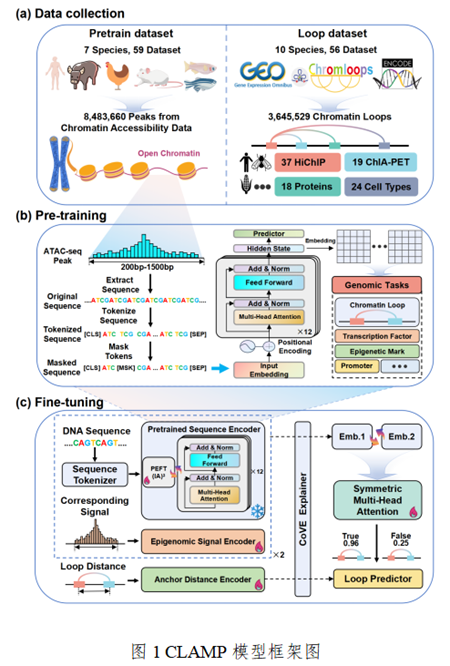
为克服深度学习模型常见的“黑箱”问题,本研究还引入了创新的可解释性分析方法——CoVE(Coefficient of Variation Explainability)。通过CoVE分析发现,基因组距离在染色质环形成中具有关键作用,但其影响高度依赖于细胞环境;相比之下,序列特征和表观遗传信号的作用则更为稳定。为便于科研人员使用,团队同步开发了用户友好的在线Web平台CLAMP+(https://clamp.sysomics.com)。
实验结果表明,在涵盖10个物种、18种介导蛋白和24种细胞类型的56个数据集中,CLAMP均表现出优越的预测性能,显著优于现有同类方法。更为突出的是,CLAMP展现出强大的跨场景泛化能力,能够基于进化上保守的序列特征,实现跨物种及跨细胞类型的染色质环精准预测。
该研究得到了国家重点研发计划、国家自然科学基金、陕西省杰出青年科学基金及北京市科技新星计划等项目的资助。
论文链接:https://link.springer.com/article/10.1186/s13059-026-03948-9
编辑:张晴
终审:刘玉峰